近日,南昌大学黄志兵教授课题组在《Food Research International》上发表了题目为“Comparative transcriptome analysis of Monascus purpureus at different fermentation times revealed candidate genes involved in exopolysaccharide biosynthesis”的研究性论文。

摘要:胞外多糖(EPS)是药用食用菌红曲霉的代谢产物,具有抗氧化、免疫调节和抗炎作用。但其生物合成机理尚不清楚,阻碍了其开发利用。本研究对紫色红曲的发酵条件进行了优化,并通过比较转录组学分析了EPS合成的机制和影响。最佳培养基组成为40g/L甘露糖,4g/L酵母粉,1 g/L MgSO4⋅7H2O, 0.8 g/L KH2PO4,1.6 g/L K2HPO4⋅3H2O, 2ml/L吐温80,最佳培养条件为接种量7%,培养温度30℃,初始pH 6.0, 180 rpm,培养4d。共获得8095个unigenes,鉴定出17种EPS合成的关键酶。有趣的是,与发酵2天相比,发酵4天组有12个碳水化合物代谢亚类富集,大部分差异表达基因(DEGs)上调,但只有9个差异表达基因(DEGs)随着发酵时间的延长而富集,所有差异表达基因(DEGs)均下调。本研究为提高EPS含量提供了理论基础,揭示了EPS合成动态,为未来EPS分子修饰和基因敲除研究提供了重要靶点。
研究内容
1、培养基对EPS产生的影响
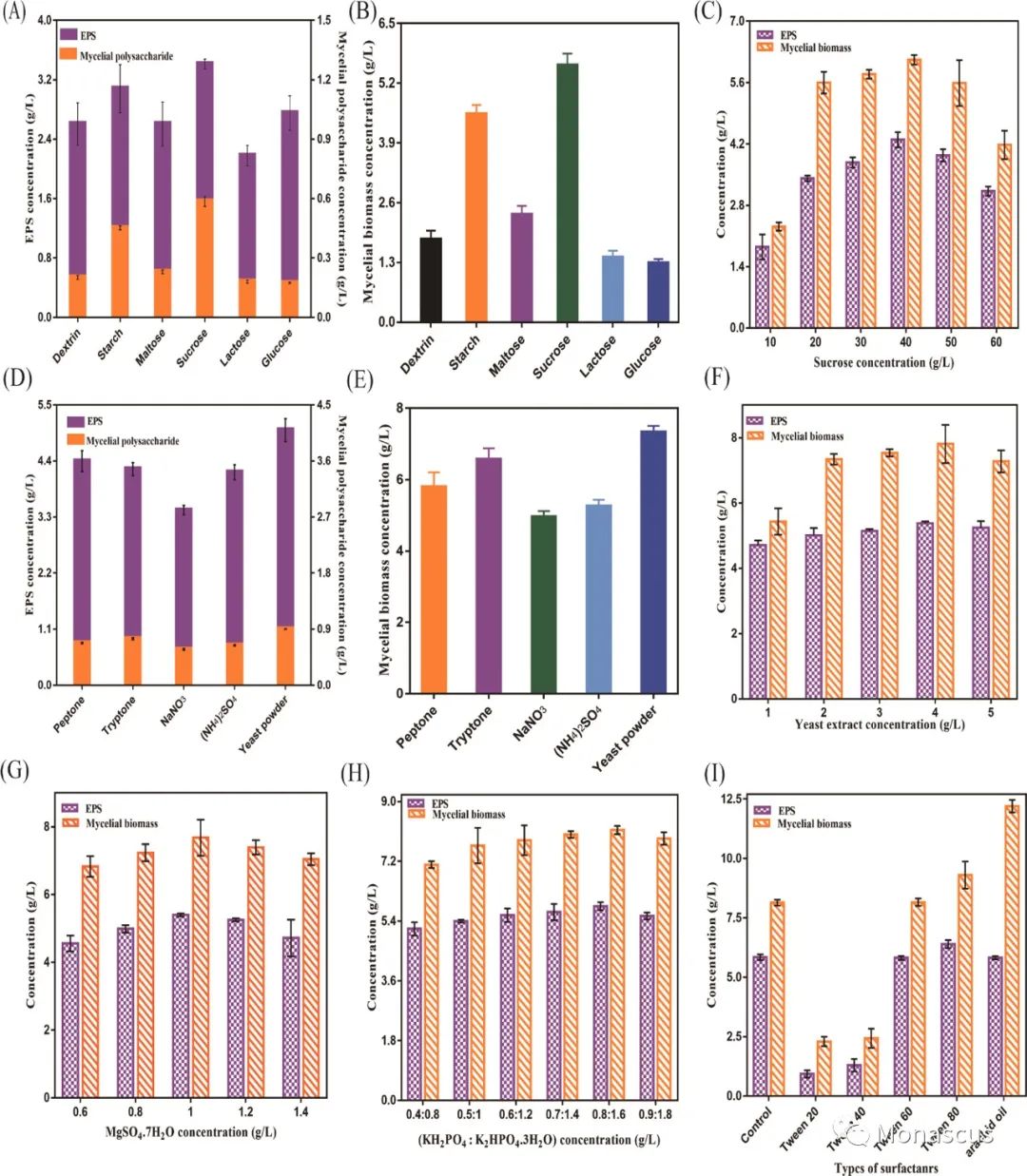
图1:紫色红曲发酵培养基的优化。不同碳源对(A)EPS和菌丝多糖产量及(B)菌丝生物量浓度的影响。(C)糖浓度对EPS产量和菌丝生物量浓度的影响。不同氮源对(D)EPS和菌丝多糖产量及(E)菌丝生物量浓度的影响。酵母提取物浓度(F)、MgSO4⋅7H2O浓度(G)、KH2PO4和K2HPO4⋅3H2O浓度(H)和表面活性剂类型(I)对EPS产量和菌丝生物量浓度的影响。
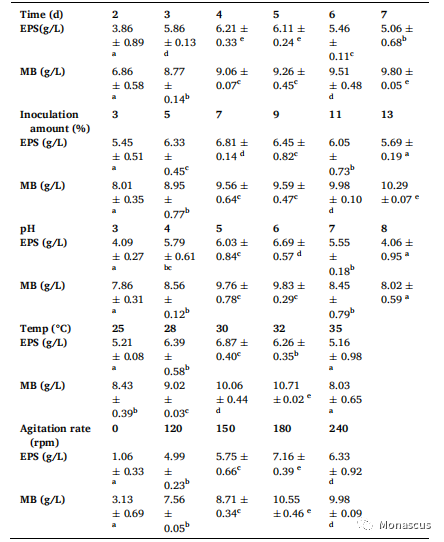
2、培养条件对EPS产量的影响
表1: 不同培养条件对红曲霉胞外多糖(EPS)及生物量的影响
3、形态分析
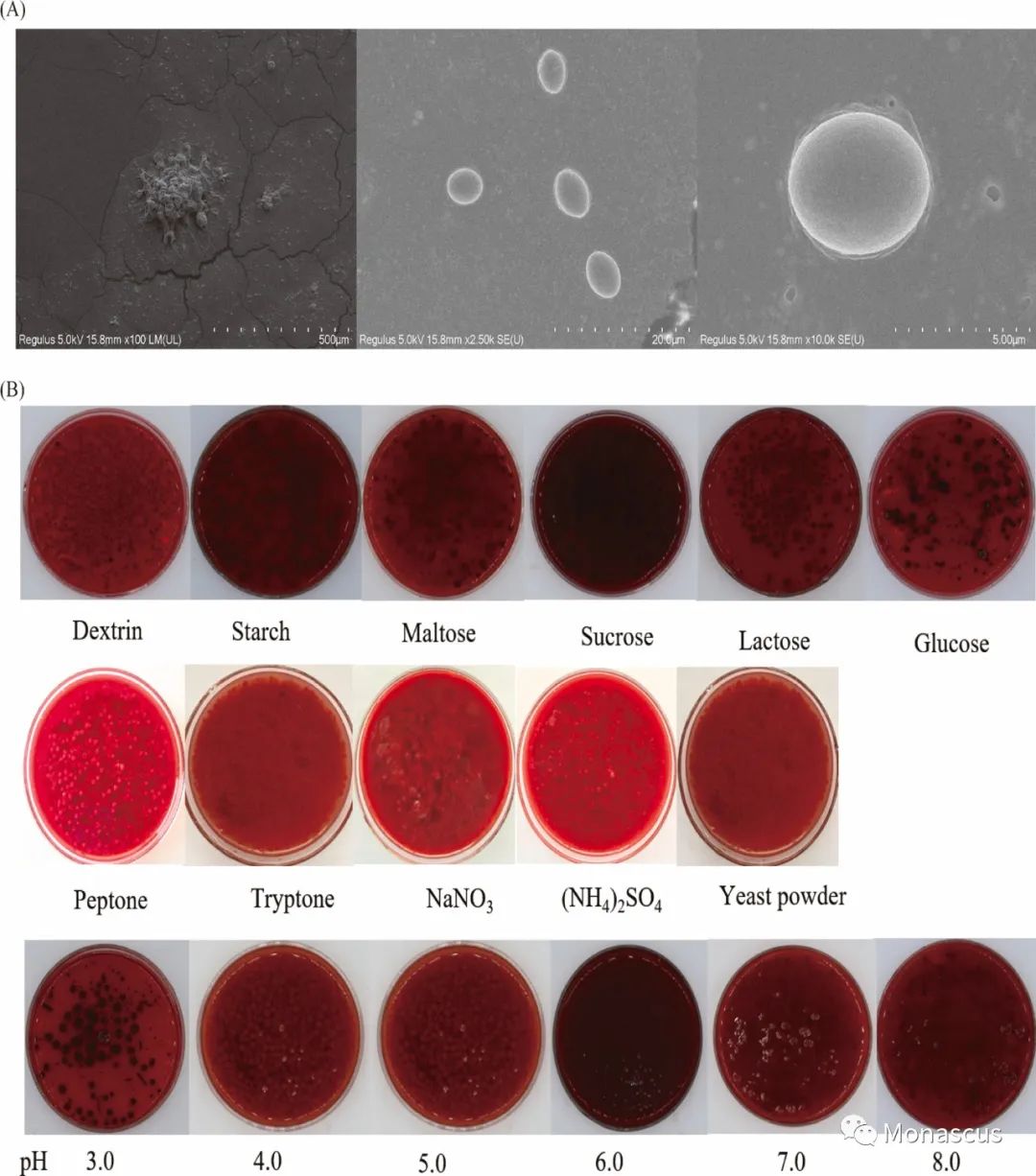
图2:(A)在100×、2500×、10000 ×放大率下,从左到右排列的紫色红曲扫描电镜图像。(B)不同发酵条件下菌丝体宏观形态。
4、紫色红曲40269不同发酵时间的RNA-seq测序
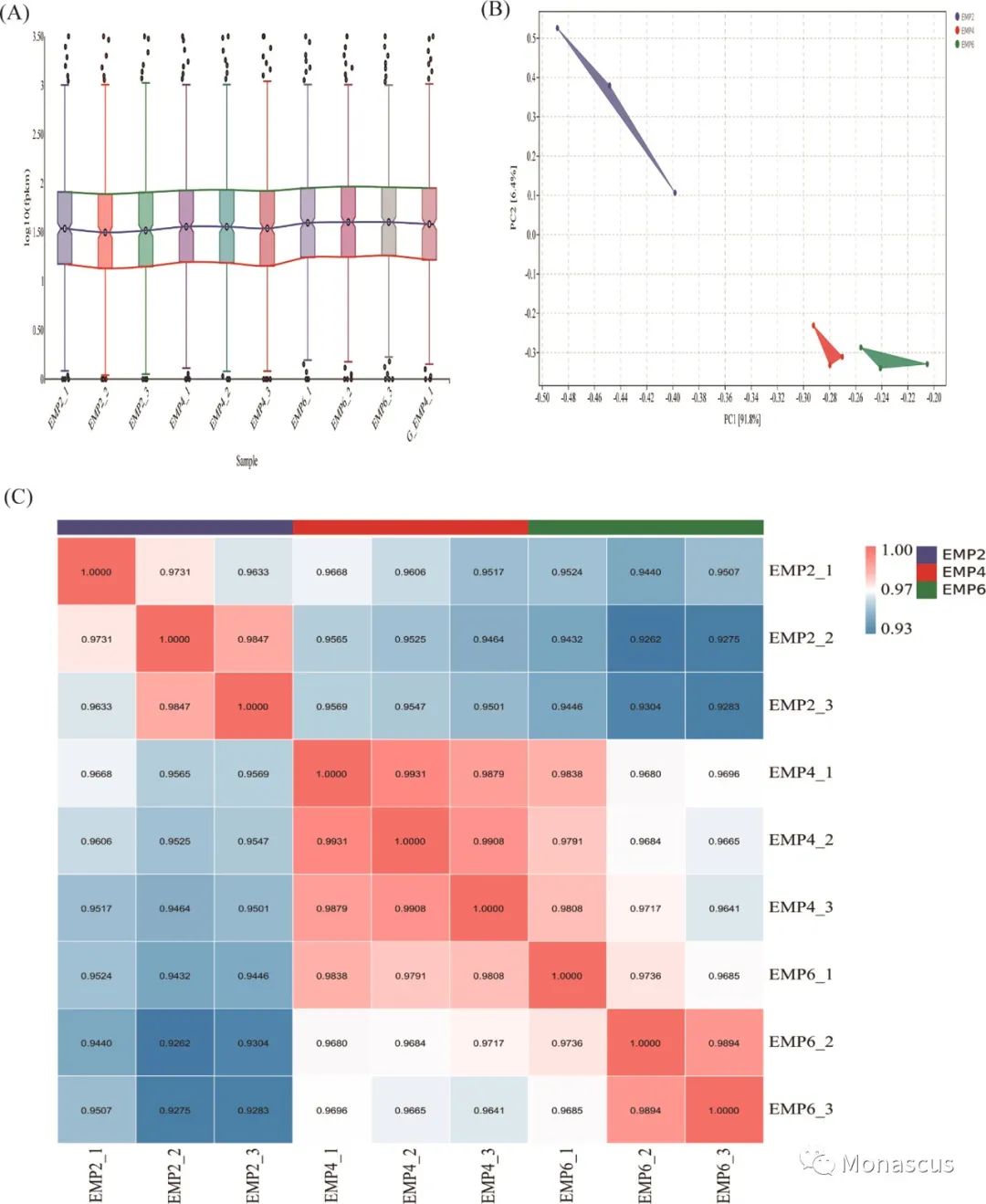
图3:用RNA-Seq分析不同发酵时间紫色红曲的基因表达谱。(A)样本表达式分布图。(B)紫色红曲在EMP2、EMP4和EMP6阶段的主成分分析(PCA)。(C)紫色红曲三个生物重复序列转录组间相关性的Spearman相关系数(SCC)分析。
5、unigenes的功能注释
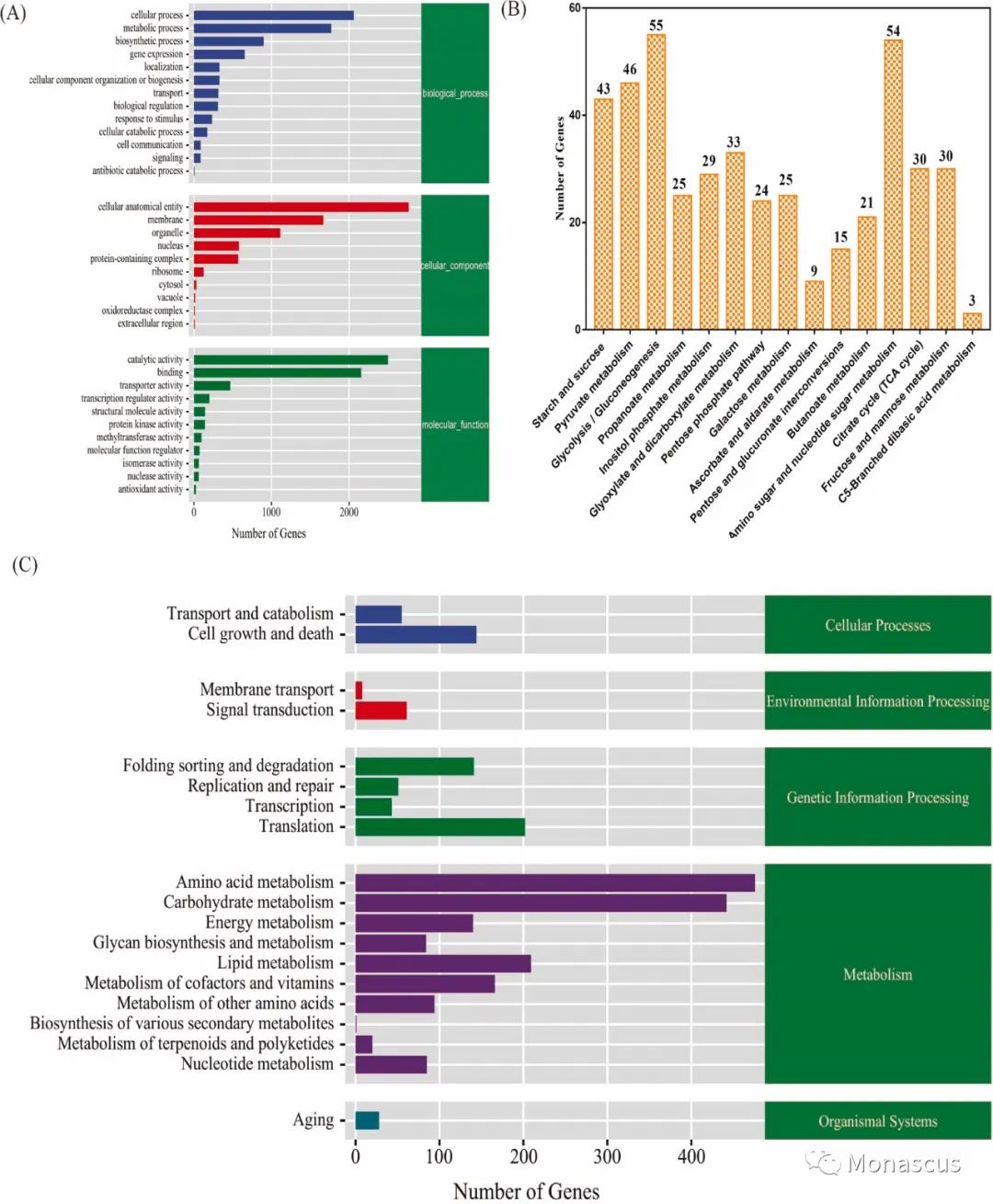
图4:紫色红曲的所有unigenes的生物信息学分析。(A) 紫色红曲的GO功能分类。(B)碳水化合物代谢途径分类。(C) KEGG通路单基因分配。
6、KEGG途径分析参与EPS生物合成的功能基因
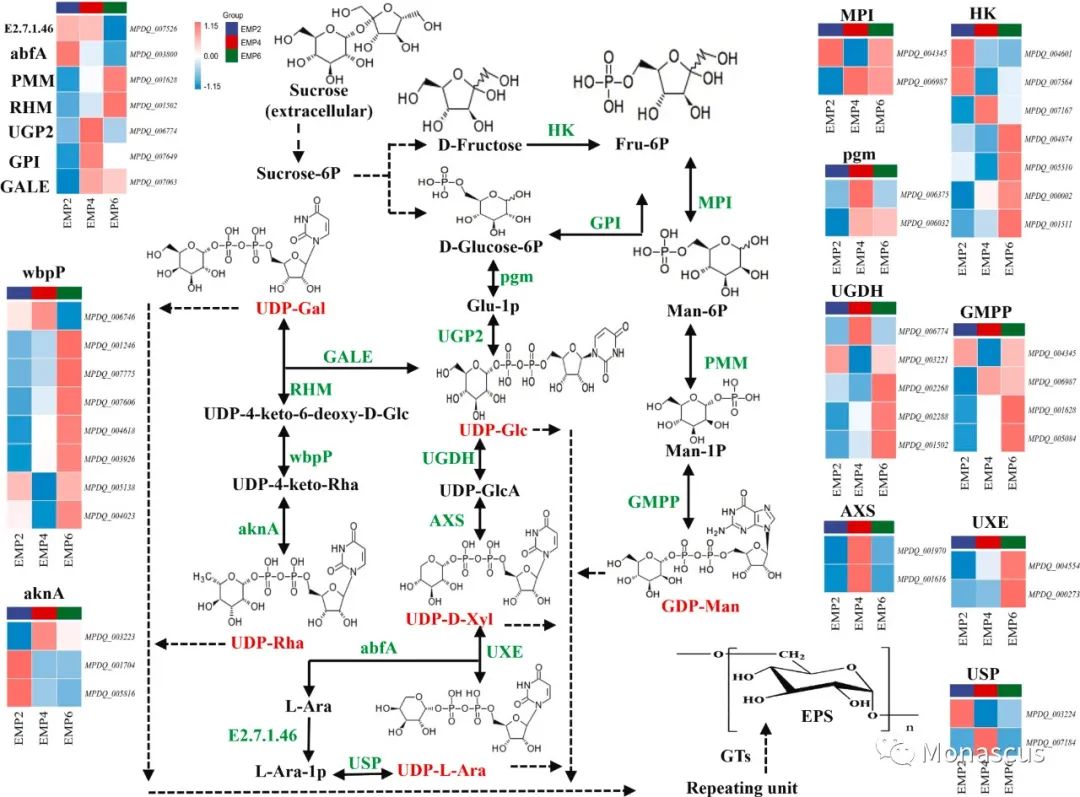
图5:紫色红曲EPS的生物合成途径。活化的单糖单位显示为红色。编码该酶的每个单基因在每一步的表达水平被显示为热图。列EMP2、EMP4、EMP6分别对应发酵时间的第2天、第4天、第6天。蓝色和红色分别代表低和高表达水平。非虚线箭头表示酶促反应,虚线箭头表示经过几个步骤的几个酶促反应。
7、发酵过程中紫色红曲40269的转录谱变化
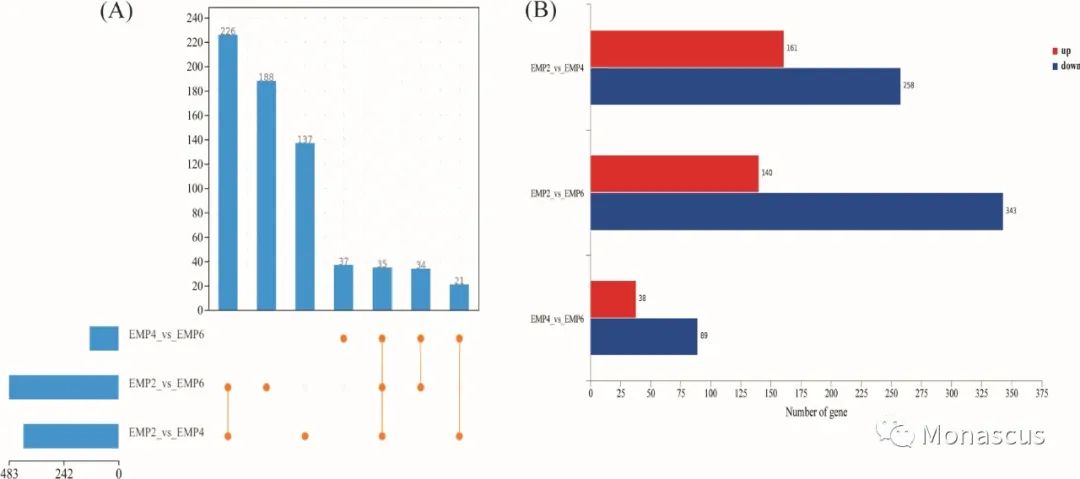
图6:紫色红曲发酵过程中DEGs的数量。(A)在EMP2、EMP4和EMP6处的共同和唯一的DEGs在加粗维恩图中表示。(B)不同组的DEGs数量。
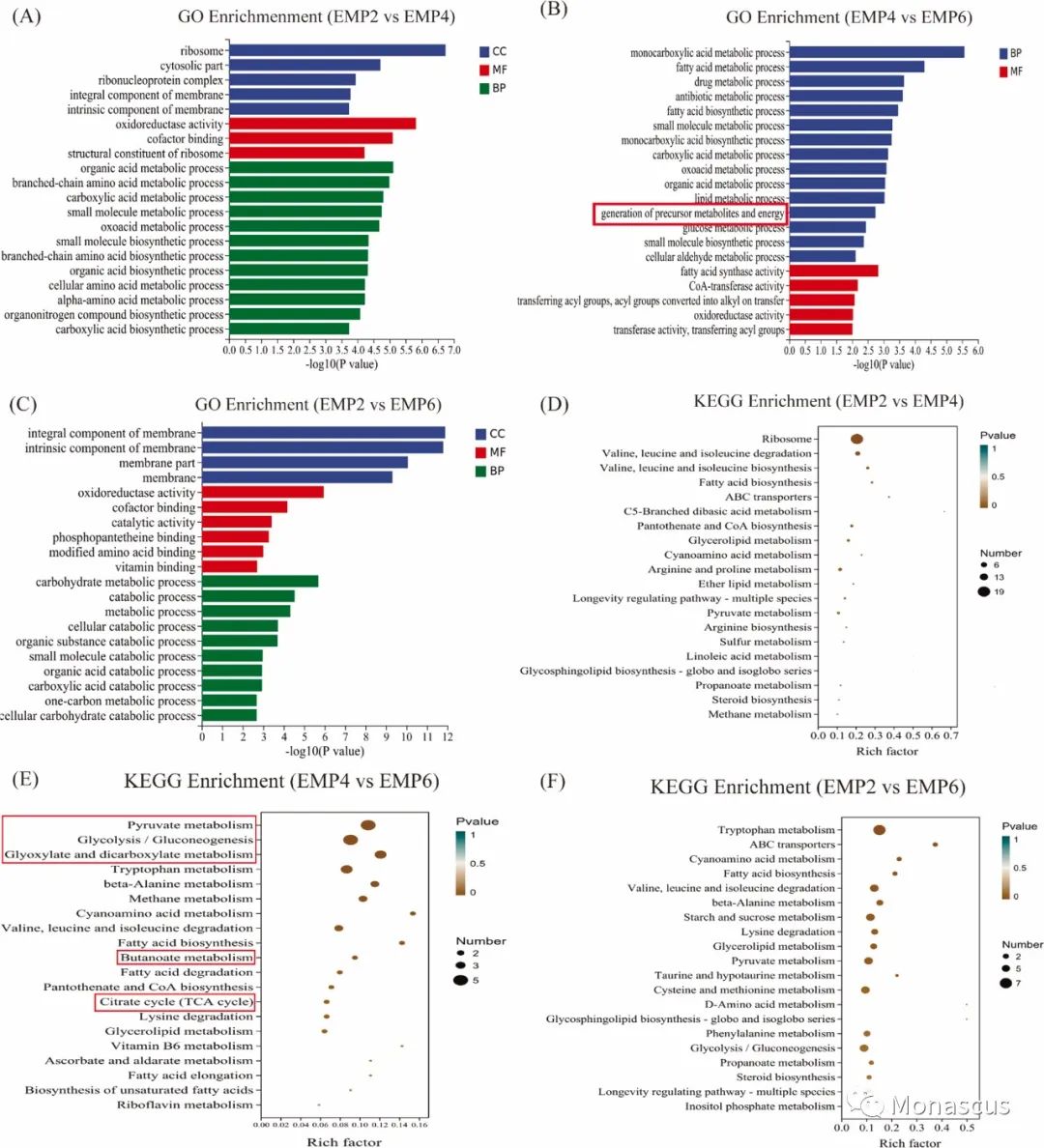
图7:DEGs的GO和KEGG富集分析。比较组中EMP2 vs EMP4 (A)、EMP4 vs EMP6 (B)、EMP2 vs EMP6 (C)前20个GO术语的分类和主要富集的显著性图。比较组中EMP2 vs EMP4 (D)、EMP4 vs EMP6 (E)、EMP2 vs EMP6 (F)前20个通路的显著性气泡图。
8、关键酶的验证和分析
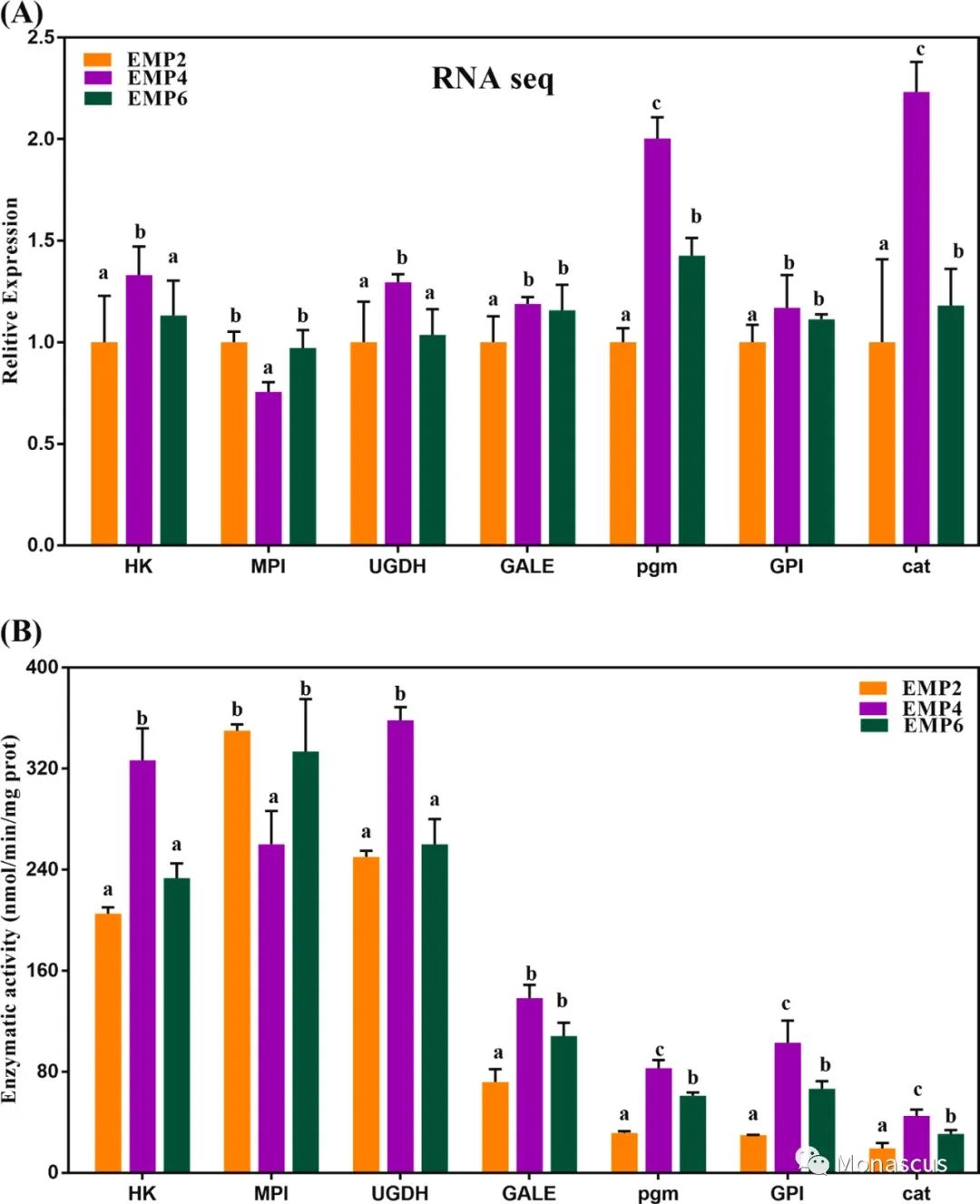
图8:(A) RNA-seq结果中影响EPS合成的关键酶(HK、MPI、UGDH、GALE、pgm、GPI和cat)的表达。(B)影响紫色红曲不同发酵时期EPS合成的关键酶(HK、MPI、UGDH、GALE、pgm、GPI和cat)的活性。
结论:
最佳培养基组成为:40 g/L甘露糖,4 g/L酵母粉,1 g/L MgSO4⋅7H2O, 0.8 g/L KH2PO4, 1.6 g/L K2HPO4⋅3H2O, 2ml /L吐温80,最佳培养条件(接种量为7%,培养温度30℃,初始pH6.0, 180 r/min,培养4 d)。
鉴定出参与EPS生物合成的17种关键酶(HK、PMM、MPI、GMPP、GPI、UGP2、pgm、AXS、abfA、GALE、E2.7.1.46、UGDH、UXE、USP、RHM、wbpP、aknA),并对其进行DEGs分析。
酶联免疫分析验证了几种关键酶,结果与RNA-seq分析结果一致。本研究阐明了EPS生物合成的可能机制,并对其合成途径进行了表征,为未来EPS分子修饰和基因敲除研究提供了重要靶点。然而,未来的研究应该集中在调控EPS合成的关键候选基因的功能。





